การฝังเพื่อนบ้านแบบเฟ้นสุ่มแจกแจง t
| ส่วนหนึ่งของเนื้อหา |
| การเรียนรู้ของเครื่อง และ การทำเหมืองข้อมูล |
|---|
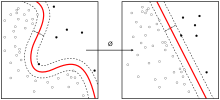 |
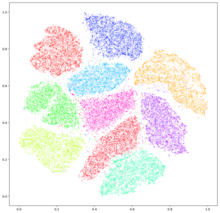
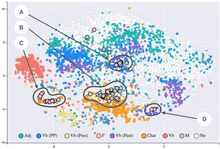
การฝังเพื่อนบ้านแบบเฟ้นสุ่มแจกแจง t (t-distributed stochastic neighbor embedding, t-SNE) เป็นวิธีการทางสถิติสำหรับการแสดงข้อมูลมิติสูงด้วยการกำหนดตำแหน่งข้อมูลแต่ละจุดในแผนที่สองมิติหรือสามมิติ โดยมีพื้นฐานจากขั้นตอนวิธีการฝังเพื่อนบ้านแบบเฟ้นสุ่มที่พัฒนาขึ้นครั้งแรกโดยเจฟฟรีย์ ฮินตัน และ แซม โรไวส์ (Sam Roweis)[1] แล้วได้รับการเสนอรูปแบบการแจกแจงที โดย เลาเรินส์ ฟัน แดร์ มาเติน (Laurens van der Maaten) และฮินตัน[2] วิธีนี้เป็นการลดมิติแบบไม่เชิงเส้น ซึ่งเหมาะสำหรับการฝังข้อมูลมิติสูงลงในพื้นที่มิติต่ำ (2 มิติ หรือ 3 มิติ) สำหรับการแสดงให้เห็นเป็นภาพ โดยเฉพาะอย่างยิ่ง เมื่อจัดเรียงชุดข้อมูลมิติสูงใน 2 หรือ 3 มิติ ชุดที่คล้ายกันจะสัมพันธ์กับความน่าจะเป็นสูงในบริเวณใกล้เคียง และชุดที่แตกต่างกันจะสัมพันธ์กันในบริเวณที่ห่างไกล
ขั้นตอนวิธี t-SNE โดยหลักแล้วประกอบด้วย 2 ขั้นตอน โดยขั้นแรก คือการสร้างการแจกแจงความน่าจะเป็นเพื่อให้คู่ของข้อมูลมิติสูงแต่ละคู่มีแนวโน้มที่จะเลือกกลุ่มที่คล้ายกัน ในขณะที่ชุดที่แตกต่างจะมีความน่าจะเป็นที่จะอยู่กลุ่มเดียวกันน้อย ขั้นตอนต่อมาคือ กำหนดการแจกแจงความน่าจะเป็นที่คล้ายกันสำหรับเซตบนแผนที่มิติต่ำ และค้นหาตำแหน่งของจุดในแผนที่มิติต่ำที่จะลดไดเวอร์เจนซ์คัลแบ็ก–ไลบ์เลอร์ระหว่างการแจกแจงทั้งสองให้เหลือน้อยที่สุด ขั้นตอนวิธีดั้งเดิมใช้ระยะทางแบบยุคลิด เป็นการวัดความคล้ายคลึงกันระหว่างจุดสองจุด แต่จำเป็นต้องแก้ไขอย่างเหมาะสมตามความจำเป็น
t-SNE ถูกนำมาใช้เพื่อแสดงภาพในการใช้งานที่หลากหลาย รวมถึงการวิจัยด้านความมั่นคงคอมพิวเตอร์[3] การวิเคราะห์ดนตรี[4] การวิจัยมะเร็ง[5] ชีวสารสนเทศศาสตร์[6] และการประมวลผลสัญญาณทางชีวการแพทย์[7] นอกจากนี้ยังมักใช้เพื่อแสดงภาพตัวแทนระดับสูงที่เรียนรู้จากโครงข่ายประสาทเทียม[8]
แม้ว่ามักจะมองเห็นกลุ่มก้อนได้ในแผนภาพ t-SNE แต่ก็จำเป็นต้องมีความเข้าใจที่ดีเกี่ยวกับพารามิเตอร์ t-SNE เนื่องจากกลุ่มก้อนที่มองเห็นอาจเปลี่ยนไปอย่างมากโดยขึ้นกับพารามิเตอร์ที่เลือก กลุ่มก้อนดังกล่าวยังสามารถปรากฏขึ้นมาได้จากข้อมูลที่ไม่ใช่กลุ่มก้อนจริง[9] นั่นคืออาจทำให้ได้กลุ่มก้อนปลอม ดังนั้นจึงอาจจำเป็นต้องค้นหาซ้ำโดยเลือกพารามิเตอร์และตรวจสอบผลลัพธ์ใหม่[10][11] t-SNE มักจะสามารถกู้คืนกลุ่มก้อนที่แยกจากกันได้ดี ได้มีการสาธิตให้เห็นถึงรูปแบบที่เรียบง่ายของรูปร่างกลุ่มสเปกตรัมโดยการเลือกพารามิเตอร์พิเศษแล้ว[12]
รายละเอียด
[แก้]สมมุติว่ามีชุดข้อมูล ตัวที่แสดงค่าหลายมิติ วัตถุประสงค์ของเราคือแสดงชุดข้อมูลนี้ในรูปของ ที่มีจำนวนมิติต่ำกว่าที่สามารถสะท้อนให้เห็นถึงลักษณะความคล้ายคลึงกันของชุดข้อมูลมิติสูง
พารามิเตอร์สำหรับ t-SNE ได้แก่ ค่าความงุนงง (perplexity) ของพารามิเตอร์ฟังก์ชันการสูญเสียและจำนวนการคำนวณวนซ้ำ ของพารามิเตอร์การปรับให้เหมาะสม, อัตราการเรียนรู้ , โมเมนตัม ฟัน แดร์ มาเติน ได้อธิบายไว้ว่าสมรรถนะของ t-SNE ไม่ค่อยขึ้นกับค่าความงุนงง โดยค่าความงุนงงที่เหมาะสมที่สุดนั้นต่างกันไปขึ้นอยู่กับข้อมูลที่ใช้ แต่โดยทั่วไปจะอยู่ระหว่าง 5 ถึง 50
ขั้นแรก เราคำนวณความคล้ายคลึงกันของแต่ละคู่สำหรับชุดข้อมูลมิติสูง ฟัน แดร์ มาเติน และ ฮินตัน ได้อธิบายว่า "ถ้าเลือกจุดข้อมูล โดยอิงตาม ให้เป็นสัดส่วนกับการแจกแจงความหนาแน่นความน่าจะเป็นแบบปรกติที่มีใจกลางอยู่ที่ แล้ว ความคล้ายคลึงกันระหว่าง กับ จะแสดงได้เป็นความน่าจะเป็นมีเงื่อนไข "[2]
โดยสำหรับจุดเดียวกันจะได้ว่า
คือค่าเบี่ยงเบนของการแจกแจงปรกติ ซึ่งอาจหาได้โดยวิธีแบ่งครึ่ง เป็นไปตามความสัมพันธ์ความงุนงงดังต่อไปนี้
ในที่นี้ คือเอนโทรปีของข้อมูล หากกระจุกกันอยู่อย่างหนาแน่นในพื้นที่แคบแล้ว จะเป็นค่าที่มีขนาดเล็ก
จากนั้น[ความน่าจะเป็นร่วม]] คำนวณได้โดยใช้สูตรต่อไปนี้
โดยในกรณี จะกลายเป็น 0 (นั่นคือ )
ให้ผลเฉลยตั้งต้น ได้จากการสุ่มตัวอย่างของการแจกแจงแบบเกาส์เซียนที่มีค่าเฉลี่ยเป็น 0
สุดท้าย ให้ทำซ้ำ T ครั้ง หาผลเฉลย ในขั้นตอนต่อไปตั้งแต่ขั้น t=1 ถึง t=T
คำนวณความคล้ายคลึงมิติต่ำสำหรับ ซึ่งเป็ผลเฉลยที่ t-1
ความน่าจะเป็นร่วมโดยใช้การแจกแจงที (การแจกแจงโคชี) โดยมีองศาเสรีเป็น 1
อย่างไรก็ตาม จะให้ค่าเป็น 0 สำหรับคู่ที่มีจุดเดียวกัน
ให้ไดเวอร์เจนซ์คัลแบ็ก–ไลบ์เลอร์สำหรับการแจกแจง P ของ และการแจกแจง Q ของ เป็นฟังก์ชันเป้าหมาย แล้วหาผลเฉลย ที่ทำให้มีค่าต่ำที่สุด
คำนวณความชันของฟังก์ชันเป้าหมายสำหรับแต่ละ i
ความชันของฟังก์ชันเป้าหมายและคำนวณหาผลเฉลย ลำดับที่ t จากคำตอบก่อนหน้า
การแสดงผลเฉลย ด้วยภาพทำให้สามารถเข้าใจกลุ่มของชุดข้อมูลที่มีมิติสูงได้
ข้อเสีย
[แก้]- ยังไม่ชัดเจนว่าจะดำเนินการลดมิติทั่วไปอย่างไร
- มีสมบัติที่ค่อนข้างเป็นเฉพาะที่ทำให้มีความอ่อนไหวต่อคำสาปของมิติข้อมูลโดยธรรมชาติ
- ฟังก์ชันเกาส์เซียนใช้ระยะทางแบบยุคลิด จึงได้รับผลจากคำสาปของมิติ และสูญเสียความสามารถในการแยกแยะข้อมูลตามระยะทางสำหรับมิติสูง จะกลายเป็นมีค่าเกือบเท่ากัน เพื่อบรรเทาปัญหานี้ จึงได้มีการเสนอวิธีการที่ระยะห่างจะถูกปรับโดยการแปลงกำลังตามขนาดเฉพาะของแต่ละจุด [13]
- ไม่รับประกันว่าฟังก์ชันเป้าหมาย t จะลู่เข้าที่ค่าต่ำสุดวงกว้าง
- แม้ว่าจะมีพารามิเตอร์และขั้นตอนวิธีเหมือนกัน ก็อาจได้ผลเฉลยที่แตกต่างกัน
อ้างอิง
[แก้]- ↑ Hinton, Geoffrey; Roweis, Sam (January 2002). Stochastic neighbor embedding (PDF). Neural Information Processing Systems.
- ↑ 2.0 2.1 van der Maaten, L.J.P.; Hinton, G.E. (Nov 2008). "Visualizing Data Using t-SNE" (PDF). Journal of Machine Learning Research. 9: 2579–2605.
- ↑ Gashi, I.; Stankovic, V.; Leita, C.; Thonnard, O. (2009). "An Experimental Study of Diversity with Off-the-shelf AntiVirus Engines". Proceedings of the IEEE International Symposium on Network Computing and Applications: 4–11.
- ↑ Hamel, P.; Eck, D. (2010). "Learning Features from Music Audio with Deep Belief Networks". Proceedings of the International Society for Music Information Retrieval Conference: 339–344.
- ↑ Jamieson, A.R.; Giger, M.L.; Drukker, K.; Lui, H.; Yuan, Y.; Bhooshan, N. (2010). "Exploring Nonlinear Feature Space Dimension Reduction and Data Representation in Breast CADx with Laplacian Eigenmaps and t-SNE". Medical Physics. 37 (1): 339–351. doi:10.1118/1.3267037. PMC 2807447. PMID 20175497.
- ↑ Wallach, I.; Liliean, R. (2009). "The Protein-Small-Molecule Database, A Non-Redundant Structural Resource for the Analysis of Protein-Ligand Binding". Bioinformatics. 25 (5): 615–620. doi:10.1093/bioinformatics/btp035. PMID 19153135.
- ↑ Birjandtalab, J.; Pouyan, M. B.; Nourani, M. (2016-02-01). Nonlinear dimension reduction for EEG-based epileptic seizure detection. 2016 IEEE-EMBS International Conference on Biomedical and Health Informatics (BHI). pp. 595–598. doi:10.1109/BHI.2016.7455968. ISBN 978-1-5090-2455-1.
- ↑ Visualizing Representations: Deep Learning and Human Beings Christopher Olah's blog, 2015
- ↑ "K-means clustering on the output of t-SNE". Cross Validated. สืบค้นเมื่อ 2019-04-06.
- ↑ Pezzotti, Nicola; Lelieveldt, Boudewijn P. F.; Maaten, Laurens van der; Hollt, Thomas; Eisemann, Elmar; Vilanova, Anna (2017-07-01). "Approximated and User Steerable tSNE for Progressive Visual Analytics". IEEE Transactions on Visualization and Computer Graphics (ภาษาอังกฤษแบบอเมริกัน). 23 (7): 1739–1752. doi:10.1109/tvcg.2016.2570755. ISSN 1077-2626. PMID 28113434.
- ↑ Wattenberg, Martin; Viégas, Fernanda; Johnson, Ian (2016-10-13). "How to Use t-SNE Effectively" (ภาษาEnglish). Distill. สืบค้นเมื่อ 2019-04-06.
{{cite web}}: CS1 maint: unrecognized language (ลิงก์) - ↑ Linderman, George C.; Steinerberger, Stefan (2017-06-08). "Clustering with t-SNE, provably". arXiv:1706.02582 [cs.LG].
- ↑ Schubert, Erich; Gertz, Michael (2017-10-04). Intrinsic t-Stochastic Neighbor Embedding for Visualization and Outlier Detection. SISAP 2017 – 10th International Conference on Similarity Search and Applications. pp. 188–203. doi:10.1007/978-3-319-68474-1_13.






























